Jak korzystać z dbGaP Controlled Access Data
07.05.2016 – 22:01Wpis ten jest dość specjalistyczny bo dotyczy osób, które mają dostęp do danych genetycznych pacjentów udostępnianych przez Narodowe Instytuty Zdrowia (NIH). Postanowiłam, również na życzenie użytkowników, takie wpisy popełniać (oczywiście wraz z popularnonaukowymi). Tutaj będziemy operować na przykładzie dostępu do genomu HeLa. Zauważyłam że z tym są problemy gdyż procedura jest niestety dość skomplikowana.
Sugerowana przeglądarka: Firefox. Zazwyczaj używam Opery i nie mieściło mi się w głowie że w XXI wieku istnieją jeszcze (poza Windows Update) strony wymagające konkretnej przeglądarki, ale w tym przypadku tak właśnie jest. I tak jest postęp gdyż jeszcze niedawno logowanie do dbGaP działało wyłącznie z Internet Explorera.
Oczywiście zakładam że wszelkie formalności związane z dostępem do danych zostały załatwione i dostaliśmy upragniony email:
“APPROVAL of your request [#41795-3] for access phs000640/HMB”
Co ciekawe, tzw. “downloadery” tak wrosły w amerykańską świadomość iż nawet NIH ich używa. Aby ściągnąć dane należy pobrać i zainstalować Asperę Connect – wtyczkę do przeglądarek opartych na Chrome. Pobrać ją można ze strony:
http://downloads.asperasoft.com/connect2/. Jeśli ktoś używa Opery albo Firefoksa to otrzyma informację iż wtyczka ta nie jest kompatybilna z tą wersją Chrome, można to zignorować i kliknąć poniżej na: “See all installers”
I tam wybrać z rozwijanego menu odpowiednią wersję. W moim przypadku jest to “v3.6.2 – Linux x86_64”. Do ściągania plików z NIH najlepiej użyć najnowszej wersji Aspery Connect (minimum to wersja 3.0.1).
Po ściągnięciu rozpakowujemy plik:
tar -zxf aspera-connect-3.6.2.117442-linux-64.tar.gz
Po czym uruchamiamy skrypt:
./aspera-connect-3.6.2.117442-linux-64.sh
Aspera Connect zostanie zainstalowana dla użytkownika, który uruchomił instalację. Następnie należy zrestartować Firefoksa. Aby upewnić się że wtyczka została zainstalowana można ponownie wejść na stronę, z której ją ściągnęliśmy, powinna się nam ukazać informacja: “You have the latest Aspera Connect. Thank you for using Aspera.”.
Po uporaniu się z Asperą możemy wrócić do dbGaP logując się na stronę:
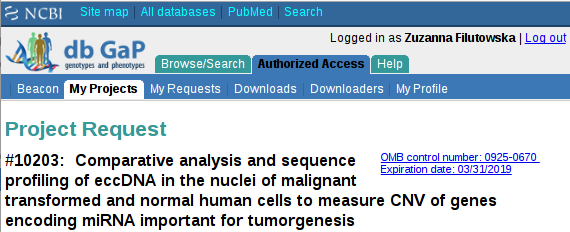
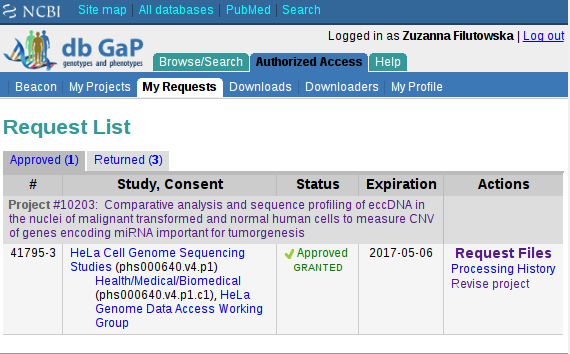
https://dbgap.ncbi.nlm.nih.gov/aa/wga.cgi?page=login. Po zalogowaniu w “My Projects” klikamy na temat naszego projektu a następnie w “My Requests”:
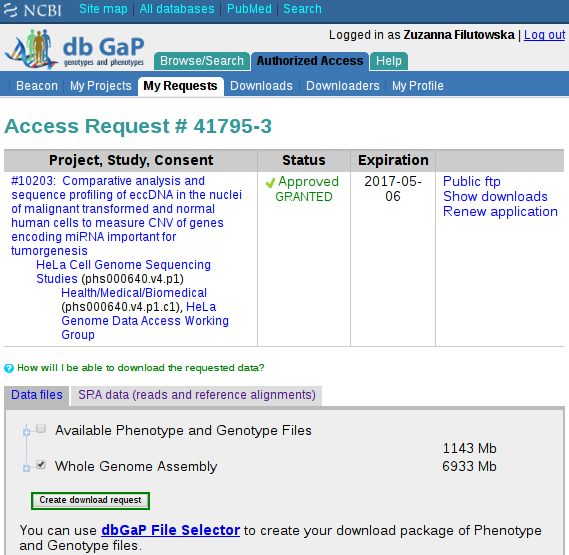
Dalej w kolumnie “Actions” klikamy na “Request Files”.
Następnie wybieramy paczkę plików, którą chcemy ściągnąć, w moim przypadku było to WGA (Whole Genome Assembly) po czym klikamy przycisk “Create download request”:
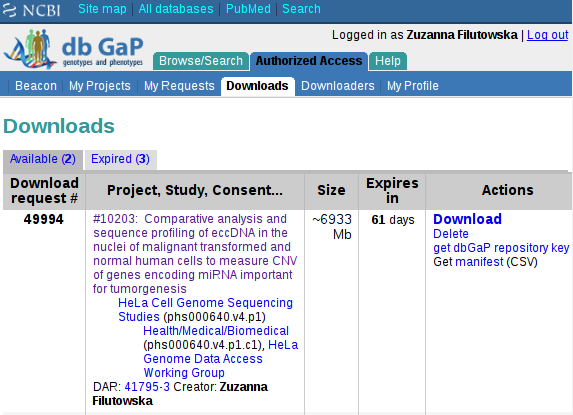
Otrzymamy informację o numerze żądania, o tym, że konieczna jest Aspera itd. Należy dłuższą chwilę zostawić stronę otwartą i poczekać na przekierowanie na stronę z listą żądań plików. Przyjdzie też emailowe potwierdzenie utworzenia żądania dostępu do plików. Po przekierowaniu klikamy na “Download”.
Ukaże się nam ponownie strona z informacją o numerze żądania oraz (jeśli Aspera jest prawidłowo zainstalowana) dwie możliwości ściągnięcia pliku przez wtyczkę oraz poniżej informacje jak ściągać pliki z linii komend bezpośrednio za pomocą ascp (Windows) lub za pomocą skryptu Perla. Skrypt Perla niestety wymaga wielu modułów Perla, których normalnie nie używam więc z niego zrezygnowałam. Klikamy więc na opcję “new directory”:
Otworzy się nam okno wyboru dowolnego katalogu (viva la liberte!) a następnie okienko Aspery. Zazwyczaj pojawia się błąd “Server aborted session: No such file or directory”. Nie przejmujemy się nim i klikamy w okienku Aspery ikonkę “Retry”. Jest to w pełni powtarzalny problem więc zakładam że to błąd oprogramowania.
Gdy pliki się ściągają możemy zacząć konfigurować narzędzia SRA, które są potrzebne by odkodować pobrane dane.
Oprogramowanie ściągamy ze strony: http://www.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=software. Używam Fedory na x86_64 więc użyłam linka w “CentOS Linux 64 bit architecture” i ściągnęłam pakiet: sratoolkit.2.6.2-centos_linux64.tar.gz
Instalacja tego oprogramowania jest bardzo prosta. Polega na rozpakowaniu pliku do dowolnego katalogu, w moim przypadku /home/platyna/Progamy:
tar -zxf sratoolkit.2.6.2-centos_linux64.tar.gz -C /home/platyna/Programy
A następnie na zmodyfikowaniu zmiennej $PATH np. przez dopisanie do ~/.bash_profile linijki:
export PATH=$PATH:$HOME/.local/bin:$HOME/bin:$HOME/Programy/sratoolkit.2.6.2-centos_linux64/bin
W tym miejscu wróćmy na chwilę na stronę z listą żądań pobrania plików (https://dbgap.ncbi.nlm.nih.gov/aa/wga.cgi?page=downloads) i ściągnijmy klucz repozytorium naszego projektu klikając na “get dbGaP repository key”. W moim przypadku plik ten to prj_10203.ngc, który umieściłam w katalogu /home/platyna/Pobrane. Plik ten zawiera hasło do konta dbGaP, niestety jest ono tam wpisane zwykłym tekstem, dlatego jeśli korzystamy ze współdzielonego systemu, np. klastra to należy zadbać o uprawnienia:
chmod 400 /home/platyna/Pobrane/prj_10203.ngc
Gdy już mamy wszystko co potrzebne możemy uruchomić konfigurator pakietu SRA wpisując na konsoli komendę:
vdb-config -i
Należy zaimportować klucz repozytorium – po wybraniu opcji “Import Repository Key” należy wskazać katalog zawierający plik ngc oraz ustawić katalogi robocze – najlepiej zostawić proponowane opcje domyślne, można też skonfigurować proxy jeśli potrzebne:
Gdy pliki się ściągną można je zdekodować używając komendy vdb-decrypt. Ja zdecydowałam się zdekodować wszystkie pliki od razu (nie zawsze jest to dobry pomysł – zwłaszcza jeśli danych jest dużo a mamy ograniczoną przestrzeń na dysku). Wykorzystałam następujący jednolinijkowiec:
for i in `find /home/platyna/Dokumenty/Badania/hela-genome/49989/ -name ‘*.ncbi_enc’`; do echo $i; vdb-decrypt –decrypt-sra-files $i; done
W ten sposób możemy cieszyć się plikami, które możemy normalnie rozpakować (z reguły są spakowane tarem lub tarem i gzipem) za pomocą komendy gzip -d albo tar -zxf. Można użyć następujących jednolinijkowców by rozpakować wszystkie pliki:
for i in `find /home/platyna/Dokumenty/Badania/hela-genome/49989/ -name ‘*.gz’`; do echo $i; gzip -d $i; done
for i in `find /home/platyna/Dokumenty/Badania/hela-genome/49989/ -name ‘*.tar’`; do echo $i; tar -xf $i; done
That’s all folks!




1 Odpowiedź to “Jak korzystać z dbGaP Controlled Access Data”
Pozdrowienia z Madrytu…
Przez jatzenty dnia 9 cze 2016